在作物进化和驯化过程中,基因组结构变异(SVs)和基因拷贝数变异(gCNVs)是重要的遗传变异来源,越来越多的证据表明SV和gCNV在调控作物表型多样性方面具有重要作用。水稻作为世界上最重要的粮食作物,同时也是植物研究的模式生物。然而,到目前为止,还未有对其SV和gCNV全面准确的鉴定报道。
2021年5月28日,四川农业大学、中科院遗传与发育研究所、河北大学等单位合作在Cell杂志发表了题为:“Pan-genome analysis based on 33 high-quality assemblies provides insights into hidden genomic variations in rice”的研究论文。该研究对33个高质量的水稻基因组进行了系统的基因组比较分析,鉴定到了大量先前尚未发现的SVs和gCNVs,探究了SV在亚洲栽培稻群体中的分布规律和形成机制,揭示了大量SVs和 gCNVs与基因表达量变化显著相关,发现了多个与重要农艺性状相关的复杂优异等位变异,证实了SV和gCNV对调控水稻农艺性状的重要作用。

黄色标记部分为河北大学杜会龙课题组参与人员。注:王淏博士为本实验室联培博士
研究人员首先选取了遗传背景具有高度代表性的33个水稻材料,包括亚洲栽培稻各亚群代表性材料和非洲栽培稻材料,以及水稻生产和育种上广泛使用的优良品种和核心亲本材料。并对其中31份材料进行了长片段测序、高质量基因组组装及基因注释。一系列基因组质量评估结果表明所有31基因组都达到了参考基因组水平。
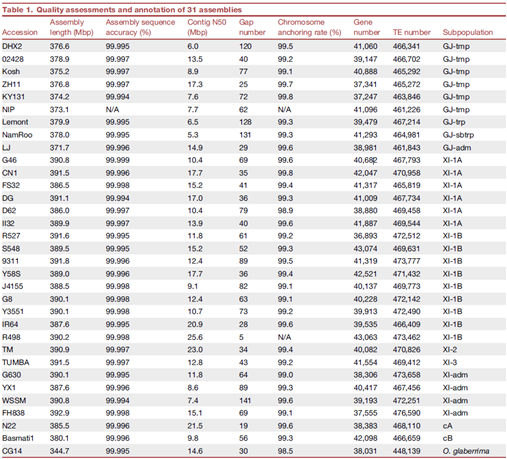
31个水稻材料的基因组组装及注释情况
利用这33个高质量水稻基因组序列,研究人员进行了系统的基因组比较分析工作,共鉴定到 171,072个相对于日本晴基因组的非冗余结构性变异(SVs),并利用了Bionano,Hi-C和PacBio reads等方法证明了其鉴定到SV的高度准确性。通过与发表的SV比较,发现其中绝大多数SV在先前研究中均未鉴定到。对SV在基因组上的分布分析发现SV在染色体上非均匀分布,共检测到140个SV热点区域。同时,研究人员利用非洲栽培稻CG14作为外群,对亚洲栽培稻群体中的SV序列的祖先型进行了推断。共明确地推断了130,862个SV基因组结构性变异的祖先型,将明确发生在亚洲栽培稻中的SVs定义为dSVs (derived-state SVs)。dSVs的推断有助于理解在亚洲栽培稻中自然选择和人工驯化作用于基因组结构性变异的方向和生物学意义。如研究人员发现:先前报道的两个与独脚金内酯合成相关基因SLB1和SLB2 被认为是在籼稻群体中缺失,导致优良性状(分蘖增加和独脚金合成减少)而被人工选择到。但结合该SV的分化状态及群体分布等分析,发现很可能是粳稻中获得SLB1和SLB2基因序列,其原因可能是其能帮助磷的吸收提高产量而被保留下来。
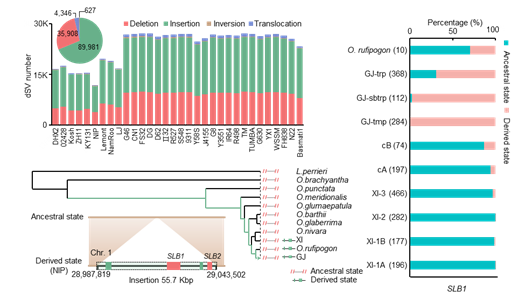
利用非洲栽培稻CG14对亚洲栽培稻群体中的SV序列的祖先型进行推断及与独脚金内酯合成相关基因SLB1和SLB2
研究人员进一步提取了蜀恢527基因组明确受到dSVs影响的基因(dSV-genes)和没有受到SVs影响的基因(nonSV-genes),并结合蜀恢527中多份不同胁迫处理和不同发育时期的转录组数据,发现dSV-genes在整体表达水平上显著低于nonSV-genes,且dSV-genes相对于nonSV-genes对环境胁迫更敏感。研究人员近一步利用33份材料苗期地上和地下转录组数据分析了SV与基因表达量的相关性,发现大量SVs与表达量显著相关,表明SVs在水稻进化和驯化过程中对基因表达模式有着广泛的影响。研究人员进一步对dSVs在亚洲栽培稻各个亚群中的分布进行研究,发现了大量dSVs被特异性地固定在一个或多个亚群中,暗示这些dSVs可能受到自然或人工选择并保留在相应的群体中。
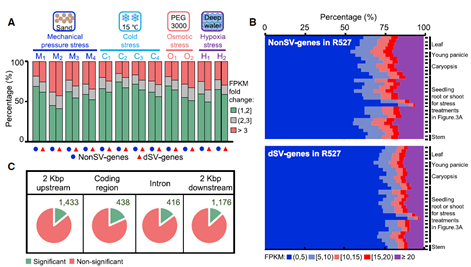
nonSV-genes和dSV-genes的基因表达情况
研究人员进一步利用33个高质量基因组序列和泛基因组中基因的蛋白序列数据,通过一系列分析,发现大量(22,549)基因在33个材料间存在基因序列拷贝数变异(gCNV)。结合33份基因组材料苗期的转录组数据,在苗期地上和地下组织中发现有很多基因的拷贝数变异和表达量之间存在显著地相关性。水稻中具有大量gCNV基因的发现,将会有助于基因拷贝数变异的产生机制和演化命运研究,也将会加快复杂区域中控制农艺性状多态性的基因组变异的挖掘和利用。如之前报道的越光中早花QTL(qDTH7-3)中含有OsMADS18两个拷贝,结合其过表达表型等分析可以很快地推断OsMADS18两个拷贝为该位点的最优候选。

33个材料中基因序列拷贝数变异(gCNV)情况
研究人员还根据SV的序列特征对其形成机制进行了分析,发现 SV主要由TEI和NHEJ机制形成,结合不同机制产生的SV的边界序列与TE的关系,发现TE除了作为转座子自身活动产生SV之外,也可以作为NAHR的底物序列驱动SVs的产生。
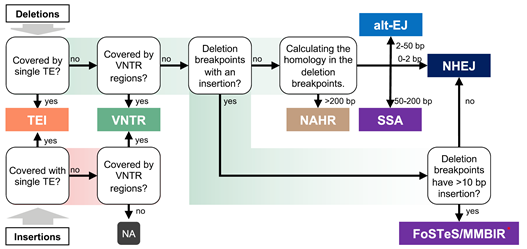
根据SV的序列特征对其形成机制进行分析
研究人员还首次构建了水稻图形基因组,结合674水稻材料的二代测序数据和图形基因组鉴定了SV,并利用群体材料的表型举例说明在研究自然变异调控农艺性状多态性方面,图形基因组和SV相较线性参考基因组和SNP有着无法代替的作用。研究人员进一步搭建了包含基因组序列和变异数据库RiceRC.Com,将会很好地方便广大研究人员使用基因组和遗传变异等数据,促进水稻功能基因组学和育种应用研究。
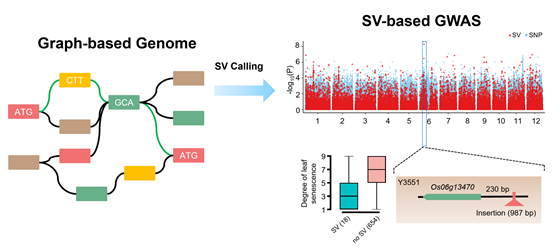
基于33份材料基因组寻找到的SV构建图形基因组并结合674水稻材料的重测序数据进行GWAS分析
这项研究成果于2021年5月28日在线发表于国际期刊Cell上。四川农业大学钦鹏教授、遗传发育所鲁宏伟博士、河北大学杜会龙教授(原遗传发育所博士)、四川农业大学王淏博士(河北大学杜会龙课题组联培博士生)、四川农业大学陈薇兰博士和遗传发育所陈倬博士为论文共同第一作者。四川农业大学教授李仕贵、遗传发育所研究员梁承志和四川农业大学教授钦鹏为论文共同通讯作者。河北大学杜会龙课题组联合培养博士生王淏为共同第一作者,工作人员何强博士、研究生张宏宇和李轩照参与了部分工作。
招贤纳士:河北大学杜会龙课题组现面向海内外招聘一名人工智能、机器学习、生物大数据分析或基因组学方向的科研人员。杜会龙,教授,河北大学高层次引进人才,以第一作者(含共同)在Cell(2020,2021,2021)、Nature Communications(2017,2019)、Molecular Plant(2017,2020)和Science China Life Sciences(2019,2020)发表多篇论文。目前课题组主要开展的研究方向是基于各类测序数据的基因组组装软件开发,复杂植物的高质量基因组构建及后续比较基因组、三维基因组调控、功能基因组分析、群体分析、基于图形基因组的泛基因组分析以及生物信息学个性化分析流程搭建等研究工作。实验室于2020年11月建成,目前已完成平台搭建、资源收集、团队建设等,实验室现有工作人员3名,博士生4名和硕士生8名。实验室目前开展的多个大型项目新颖、有趣、有意义且有挑战,欢迎有志之士加盟。目前实验室已完成多个植物材料的高质量基因组构建、比较基因组、进化、群体分析及三维基因组调控等相关分析。目前还需要一名人工智能、机器学习、生物大数据分析或基因组学方向的科研人员,涉及图形基因组算法开发、基于多组学的信息流算法开发和完成100多个处于不同进化地位的物种基因组分析及模型构建等研究工作,同时实验室已与计算机学院达成初步合作事宜,欢迎有计算机背景的人才加入!!!如有意向,请将简历发送至genomebioinfo@163.com。