燕麦(Avena sativa L., 2n=6x=42, AACCDD)作为全球重要的粮饲兼用型作物,不仅是人类的优质营养来源,同时也是牲畜的优质饲料,在保障全球粮食安全和缓解畜牧业饲料短缺问题中发挥着重要作用。燕麦属包含约30个野生种,蕴藏了丰富的优异基因资源,对于栽培燕麦遗传改良和胁迫抗性能力提升具有重要意义。其中,裂稃燕麦(Avena barbata,2n=4x=28,AABB)因其具有广泛的环境适应性而成为了研究植物生态适应机制和物种适应性演化的重要模型物种。此外,相较于其他燕麦野生种及栽培种,裂稃燕麦表现出了对各类主流燕麦病害如黑穗病、红叶病、锈病、白粉病等的广谱抗性。同时,裂稃燕麦基因组(AABB)中含有相对于栽培燕麦基因组(AACCDD)所特殊的B亚基因组。因此,解析裂稃燕麦基因组信息,对于揭示燕麦属亚基因组演化以及挖掘与环境适应相关的优异基因资源从而加速栽培燕麦的遗传改良和分子育种进程具有重要意义。
近日,JIPB在线发表了河北大学杜会龙教授团队题为“High-quality genome of allotetraploid Avena barbata provides insights into the origin and evolution of B-subgenome in Avena”的研究论文。该研究获得了燕麦属B亚基因组的完整参考基因组序列;联合基因组共线性、燕麦属各野生种物种的重测序数据、核基因组和质体基因组的系统发育以及kmer等分析,揭示了燕麦属A、B、C和D亚基因组的起源演化轨迹及其多倍化过程中的复杂网状演化模式;明确了裂稃燕麦A亚基因组的祖先供体是Avena hirtula,而B亚基因组的祖先供体则可能来自一个与Avena wiestii亲缘关系密切且可能已灭绝的二倍体物种,该发现进一步扩展了研究人员对于燕麦属多倍体物种形成及演化的认识。
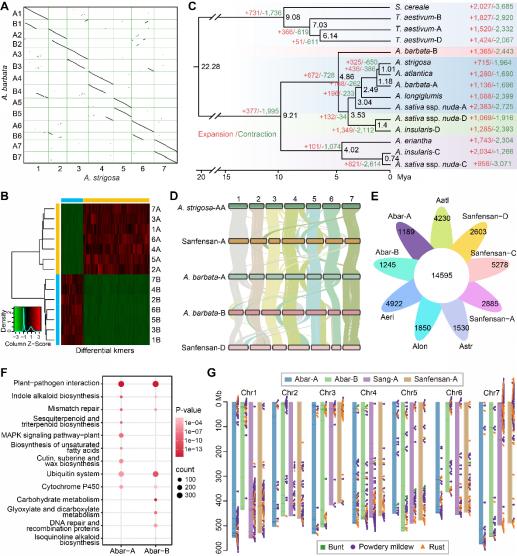
图1. 基因组进化及抗病相关基因鉴定
同时,该研究基于比较基因组学、群体遗传学和干旱胁迫下的组学等分析,在裂稃燕麦基因组尤其是B亚基因组上鉴定到了大量与抗病性(白粉病、锈病和黑穗病)和干旱胁迫响应相关的基因,阐明了裂稃燕麦强环境适应力的基因组学基础,为进一步提升栽培燕麦的干旱与病虫害抗性提供了重要基因资源。
总之,该研究填补了燕麦属B亚基因组研究的空白,揭示了燕麦属物种基因组的演化轨迹,并挖掘到了大量与环境适应相关的优异基因资源,为推动燕麦遗传改良和分子设计育种提供了重要支撑。
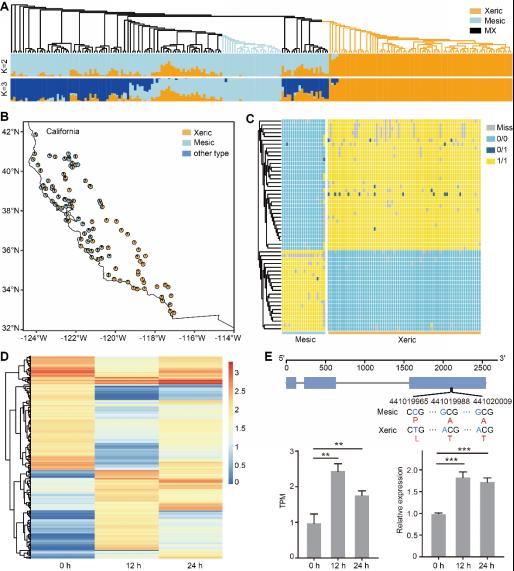
图2. 裂稃燕麦群体分析及环境适应性相关基因的鉴定
河北大学生命科学学院青年教师何强、硕士研究生肖瑶为该论文共同第一作者,河北大学杜会龙教授为论文的通讯作者。河北大学巩志忠教授对本研究给予了指导。该研究得到国家自然科学基金、中国科协青年人才托举工程和河北省自然科学基金等项目的资助。
论文信息:Qiang He#, Yao Xiao#, Tao Li, Yaru Wang, Yitao Wang, Yu Wang, Wei Li, Ningkun Liu, Zhizhong Gong, Huilong Du*. High-quality genome of allotetraploid Avena barbata provides insights into the origin and evolution of B subgenome in Avena [J]. Journal of Integrative Plant Biology 2025 DOI: 10.1111/jipb.13902.
https://www.jipb.net/EN/10.1111/jipb.13902
河北大学杜会龙教授团队围绕“燕麦基因组学与设计育种”,在全球燕麦种质资源收集与多年多点的表型评价,高产、优质、耐逆燕麦种质资源筛选与创制,基因组学大数据分析与高效育种平台搭建,重要农艺性状相关基因挖掘、利用与机制解析等方面开展了系统研究并取得了阶段性进展:1)栽培燕麦方面:团队通过广泛收集全球栽培燕麦种质资源,结合多年多点表型调查与多组学技术,构建了涵盖基因组、变异组、转录组、表型组、候选基因等组学信息的燕麦多组学综合数据库OatOmics,并提供了高效全方位的多组学在线分析工具,为燕麦功能基因组学和分子育种等研究提供了全面的共享平台(Li et al., 2025, Molecular Plant);同时,获得了120余份高产、优质、耐盐碱和耐干旱的优异燕麦种质资源,筛选出了一批调控燕麦产量、品质、抗病、耐逆等相关的优异等位基因,揭示了AsDOF25-AsHSFA2c-AsAGO1分子模块在调控燕麦生长发育与干旱胁迫响应平衡中的重要作用(Liu et al., 2025, Communications Biology),并联合基因组预测等大数据分析模型与效应评价等信息开展了大量杂交组合。2)在野生燕麦种质资源收集、开发、利用及远缘杂交等研究中,团队不仅阐明了燕麦属各野生种的起源、演化轨迹和基因组进化历程,同时挖掘到了大量与燕麦驯化和改良相关的重要基因组进化事件及优异基因,为燕麦新品种改良和种质创新等提供了重要指导(He et al., 2024, Nature Plants; Zhang et al., Accept in principle, Nature Genetics)。这些研究对于加速燕麦种质资源的精准鉴定、优良基因的挖掘与利用、基因功能解析、重要农艺性状的遗传改良以及分子设计育种等具有重要意义。
1. He, Q., Li, W., Miao, Y., Wang, Y., Liu, N., Liu, J., Li, T., Xiao, Y., Zhang, H., Wang, Y., et al. The near-complete genome assembly of hexaploid wild oat reveals its genome evolution and divergence with cultivated oats. Nature Plants, 2024, 10(12):2062-2078.
2. Li, W., Wang, Y., Liu, J., He, Q., Zhou, Y., Li, M., Liu, N., Liang, H., Yun, Y., Gong, Z., et al. A gap-free complete genome assembly of oat and OatOmics, a multi-omics database. Molecular Plant, 2025, 18(2):179-182.
3. Liu, N., Li, W., Qin, Y., Yun, Y., Yan, J., Sun, Q., Du, C., He, Q., Wang, S., Gong, Z., et al. Comprehensive co-expression network reveals the fine-tuning of AsHSFA2c in balancing drought tolerance and growth in oat. Communications Biology, 2025, 8:393.